
Das Medlinesystem wird links unten aufgerufen.

Die Hauptfunktionen des Systems sind Nr. 1und Nr.2 sowie 'Citations by 1 Letter...'
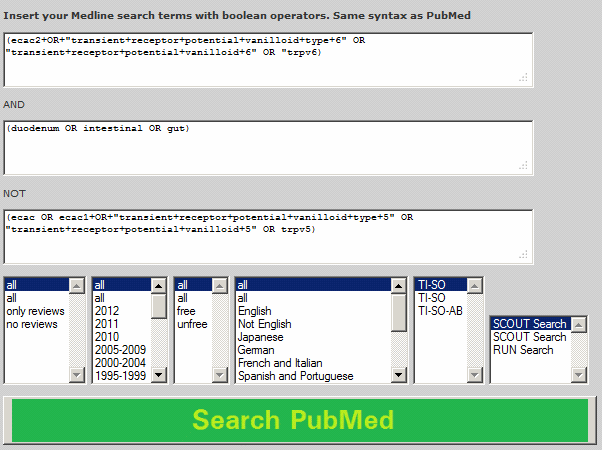
Der Suchterm wird hier eingegeben. Zur Übersichtlichkeit sind mehrere Eingabeboxen vorhanden.
Die Suche wird durch SCOUT Search auf PubMed orientierend durchgeführt und mit RUN Search auf meiner Datenbank und NCBI duchgeführt.
Filter für Reviews, Zeitraum und Sprache können verwendet werden.
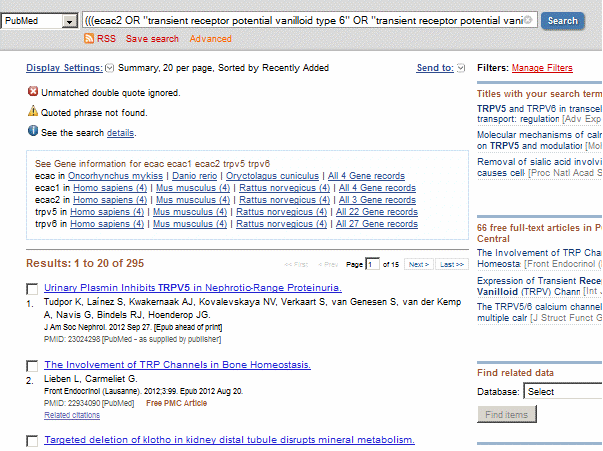
Um die Suche im gewohnten PubMed System zu prüfen, erfolgt eine Verlinkung durch Übermittelung des Suchterms.

Termübersetzung (PubMed), Ergebnismenge und ursprüngliche Suchstrategie werden vollständigdokumentiert. Vgl. PubMed-Seite !

PubNeph Papers sind in meiner Datenbank, nicht aber im PubMed enthalten.
Free Papers sind gemäß PubMed frei verfügbar. (AND free full text[sb])
Beyond PubMed seien unfrei, viele sind aber verfügbar(NOT free full text[sb])
Der Link öffnet die 'Common Linkout box', die Kreissymbole geben den Zugänglichkeitsstatus des Papers an (monatsgenau), der Barcode entspricht der PMID/UID Nummer.
MTB zeigt, daß das Paper in meinem Metatextbook aufgenommen ist.

Grün = Open Access Journal
Gelb = Hybrid Access Journal
Weiß = (meist)Authors Abstract PMC
Blau = Deutsche Nationallizenz für Privatnutzer (Freier Literaturzugang für Menschen mit Wohnsitz in Deutschland, eine Zugehörigkeit zu einer Großforschungseinrichtung ist nicht erforderlich)
Nichts = keine explizite Information
Gelb-grüner Pfeil = Frei zugängliches Paper (Editors choice, Autorenfinanzierte Publikation) durch Verlagsentscheidung

Mein System berechnet monatsgenau, ob ein Paper frei verfügbar ist.
Dieses Journal zeigt verschiedene Buttons.
Die Zahl besagt, daß das Paper in 1 Monat frei sein wird.
Mein System gibt für viele Papers Barriere-informationen, die PubMed nicht liefern kann; unsere Nationallizenz (Privatnutzer) ist ausschließlich mit meinem System prüfbar !
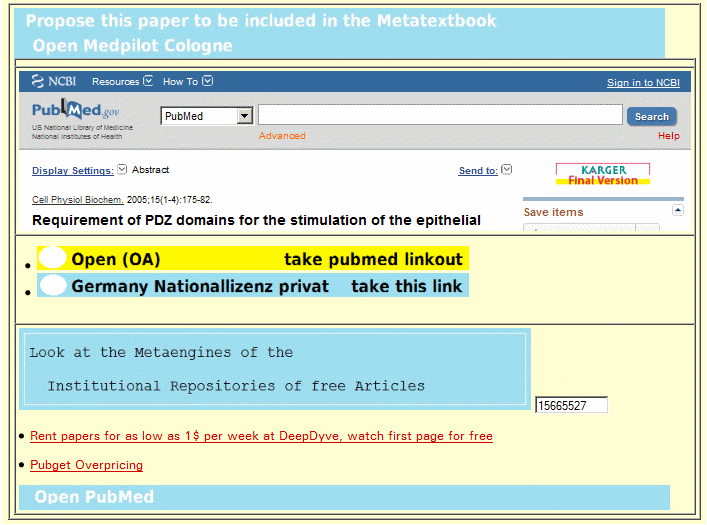
PubMed
Ausschnitt eingeframt, diverse Auslinks wie beschrieben
Die 'Common linkout box' öffnet das Dokument auf Wunsch in verschiedenen Datenbanken.
- Medpilot Cologne (Köln) zur Signaturermittlung und zum Öffnen des online Zeitschrfitenarchivs und zur Dokumentenlieferung (ca. 9 Euro)
Eingeframt ein PubMed-Ausschnitt zum artikelgenauen PDF Zugriff
wie bekannt
- Die Linkinformationen aus meinen Datenbanken, monatsgenau, weisen ins Zeitschriftenarchiv
- 4 Suchmaschinen für institutsgehostete Freiversionen und G Scholar werden geöffnet
- Auf DeepDyve kann ein Artikel für 1-4 Euro angeguckt werden
- PubGet für Nutzer, deren Bibliotheken darüber erschlossen sind (dort bitte nichts kaufen)

ausgelinkte
Ressource Verlagsseite (Beispiel Karger)
So öffnet sich ein PDF über den bekannten Pubmed Link
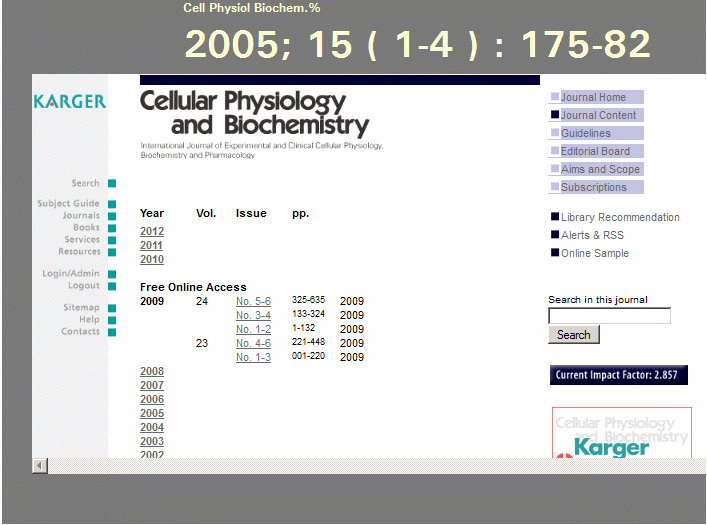
eingeframte
Ressource Verlagsseite (Beispiel Karger)
So öffnet sich ein geframtes Zeitschrfitenarchiv, man muß sich die Zitation nicht merken.
Manche Verlage möchten nicht geframt werden.
eingeframte
Ressource Deutsche Nationallizenz privat
Hier geht es zum Zeitschriftenarchiv über den DNL Proxy.
Zeitschriften freundlich gesonnener internationaler Verlage (z.B. Wiley, LWW, Informa, SAGE, Thieme, Karger) sind hier lesbar.
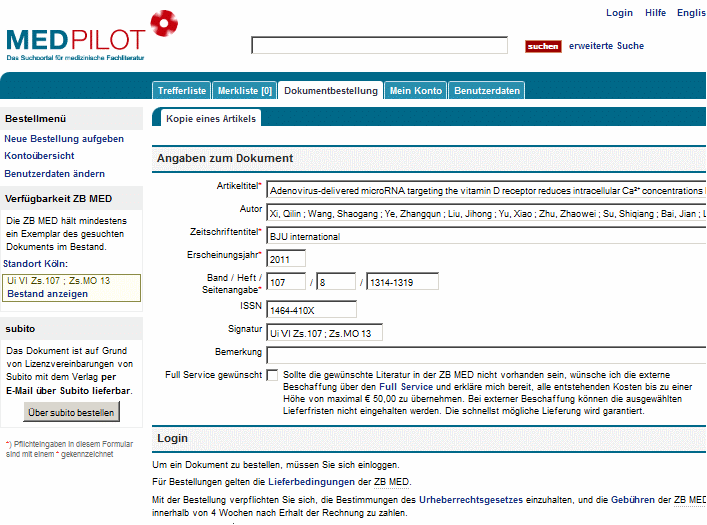
ausgelinkte
Ressource Medpilot.de
Die Metadaten können an das Medpilotsystem geschickt werden.
Es ermittelt die Kölner Signaturen, es verlinkt zu weiteren Zeitschriftenarchiven und man kann dort auch gleich ...
etwas Gescanntes bestellen, es ist aber etwas teurer als subito.
Cave: Deren PubMed ist unzureichend (Personal communication)
ausgelinkte
Ressourcen Base, Worldcat, Sci.Com., Humboldt Berlin, G Scholar
Meistens finden die Suchmaschinen, die die Institutsarchive spidern, nichts, oft sind es Pay-per-view Links, weil diese nicht mehr als Archive für Dokumente, sondern zunehmend repräsentativ verwendet werden.
Damit man sich nicht überarbeiten muß, gehen alle gleichzeitig incl G Scholar auf. Es ist aber regelmäßig enttäuschend.
ausgelinkte
Ressource deepdyve
Diese Ressource ermöglicht es, ein Paper für 1-4 Euro anzugucken.
Nimmt auch gutes Geld für freie Artikel.
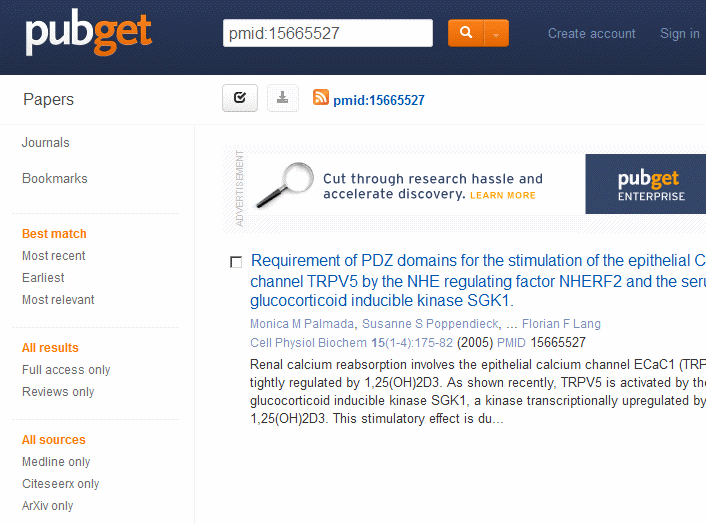
ausgelinkte
Ressource pubget
Hier wird man ausgenommen wie eine Martinsgans, wenn man auf Pay drückt: 47 Dollar für einen Hybridoffenen ist der Normalpreis.
Es ist als grundlegende Modellressource zur Fragestellung Open URL zu betrachten. Einzige deutsche angeschlossene Bibliothek ist die Stiftung Tierärztliche Hochschule Hannover. In Amerika ist es leider relevant.
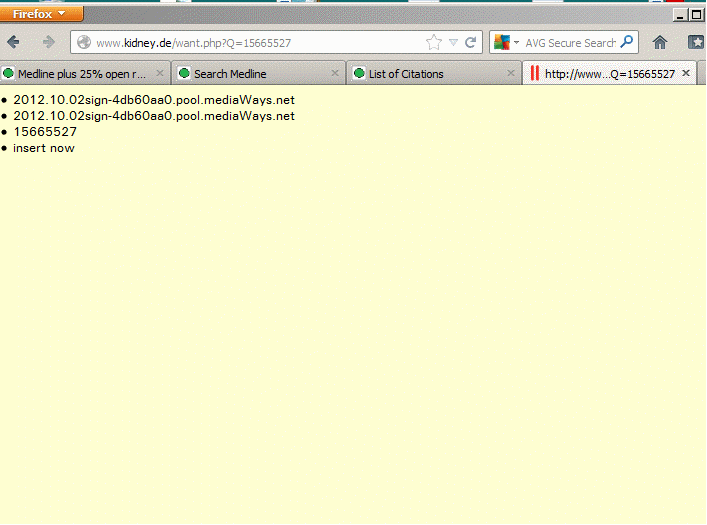
Meine Systeme sind miteinander vernetzt. So kann man mir mitteilen, daß ich ein Paper in mein Metatextbook aufnehmen soll.
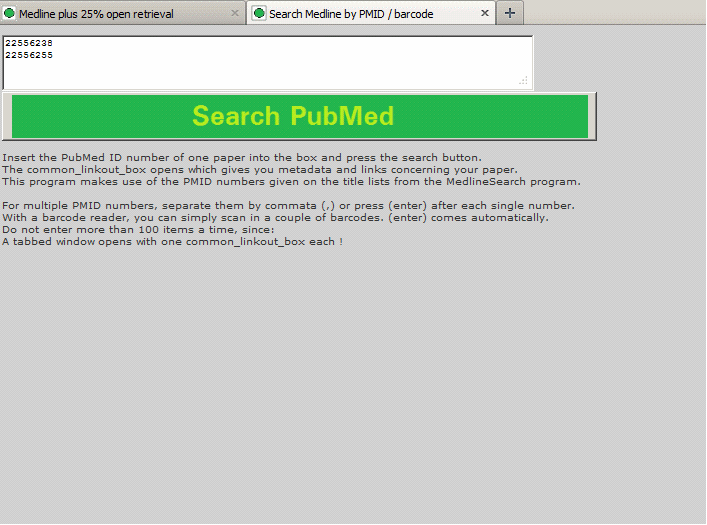
Die 'Common linkout box' öffnet sich, wenn man in der Ergebnisliste auf einen Link drückt.
Das Besondere an meinem System ist aber, daß es normal-umfangreiche Recherchen ermöglicht und erkennt, daß eine kleine Recherche mit 4000 Ergebnissen monitorbasiert nicht fehlerfrei durchgesehen werden kann und auf die ausgedruckte Liste setzt.
Die Artikel werden über ihre PubMed Nummer identifiziert (bzw. meine mit N beginnenden PubNeph Nummern), welche eingetippt oder mit einem Barcodreader eingescannt wird.
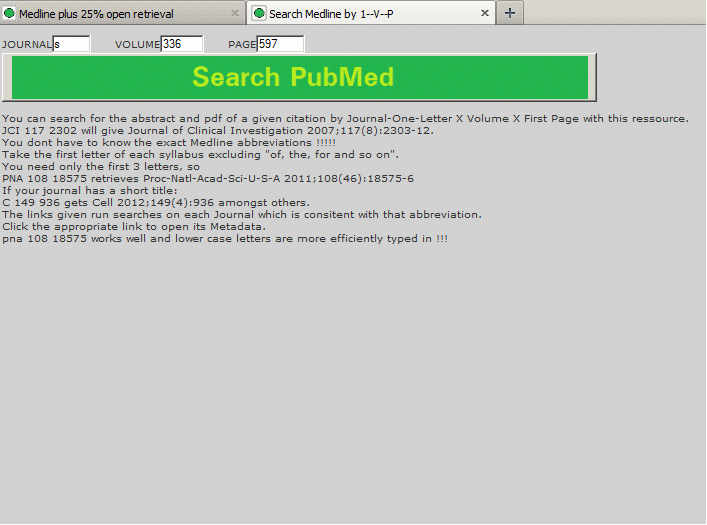
Um einen Artikel anhand seiner Zitation aufzurufen, habe ich eine Ressource geschaffen, mit welcher der auf Einzelbuchstaben abgekürzte Zeitschriftenname, Seite und Band zur PMID transformiert als Common linkout box abgebildet werden.

Alle diese Links fragen PubMed:
Science[JOUR] AND 366[VOL] AND 597[PAGE].
Man muß die Zeitschriften-abkürzungen nicht kennen (z.B. Proc-Natl-Acad-Sci-U-S-A), und nur die ersten drei Silben angeben (PNA).

Unten links ist das gesuchte Paper.
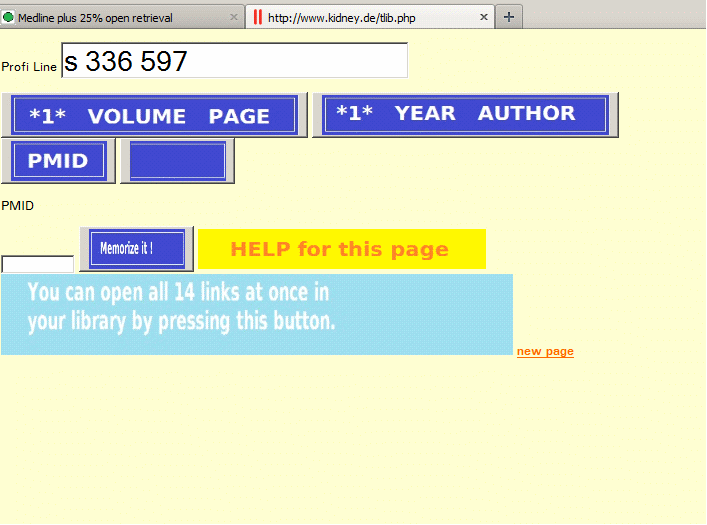
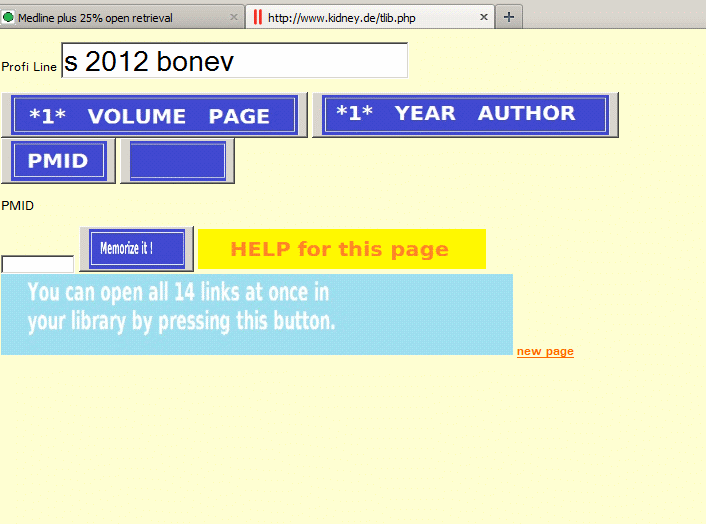
Dieses Programm tut genau dasselbe mit dem Unterschied, daß die Felder 1Letter, Vol und Page in eine Zeile geschrieben werden (Space/Komma/Punkt sind als Separatoren vorgesehen).
Man drückt dann auf den passenden Knopf, auch möglich ist die Kombination 1Letter, Jahr, Autor (irgendeiner der Autoren ohne Initialen)
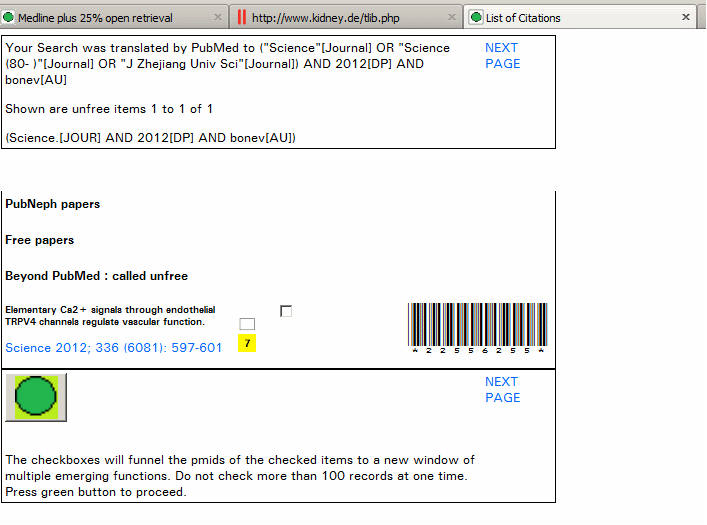
/---Suche
der Webadresse des PDF----/---Memorize it----/
Papers, die nicht frei verfügbar sind, sind oft in der Bibiothek auf USB kopierbar.
Also sollte man zu Hause versuchen, diese zu öffnen - 5% sind unerwartet frei - den Rest bearbeitet man so:
Wir benötigen eine Zusatzinstanz des tlib-Programmes, entweder in einem anderen Browser oder als 'neues Fenster' geöffnet im rechten Bildschirmdrittel, und irgendeine Zitationsquelle in den linken Bildschirmdrittel.
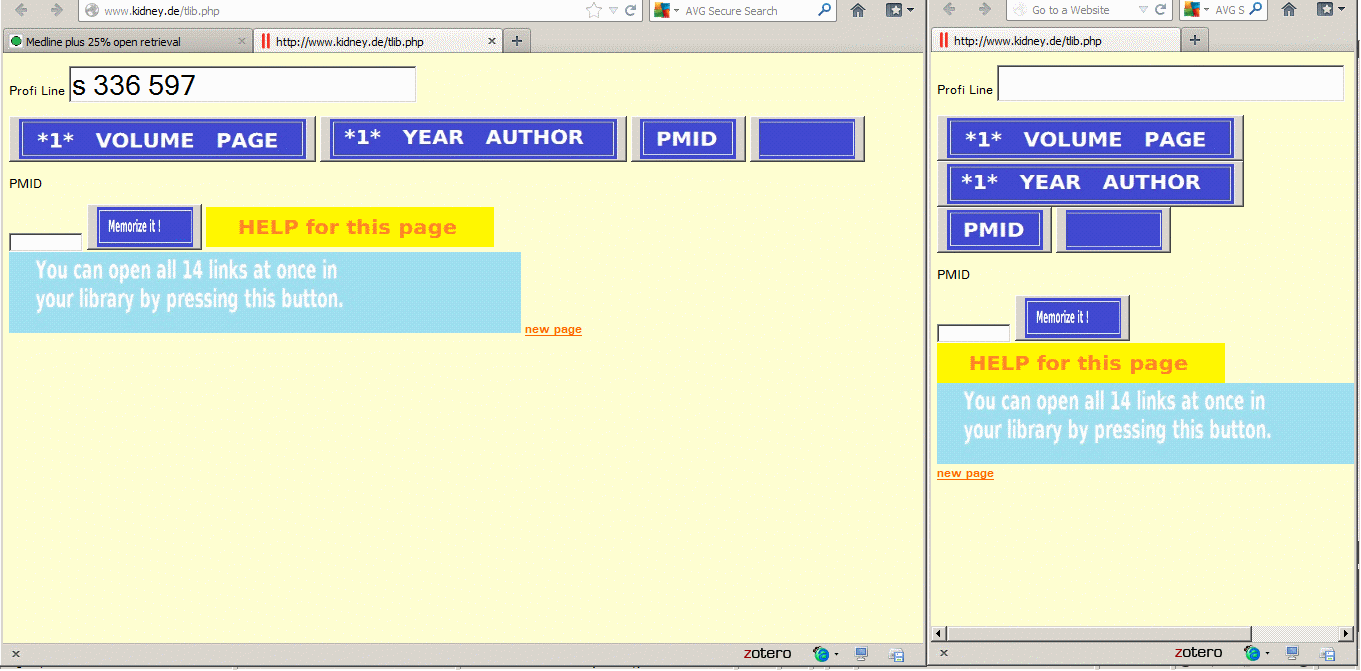
Wir geben unsere bekannte Zitation ein...
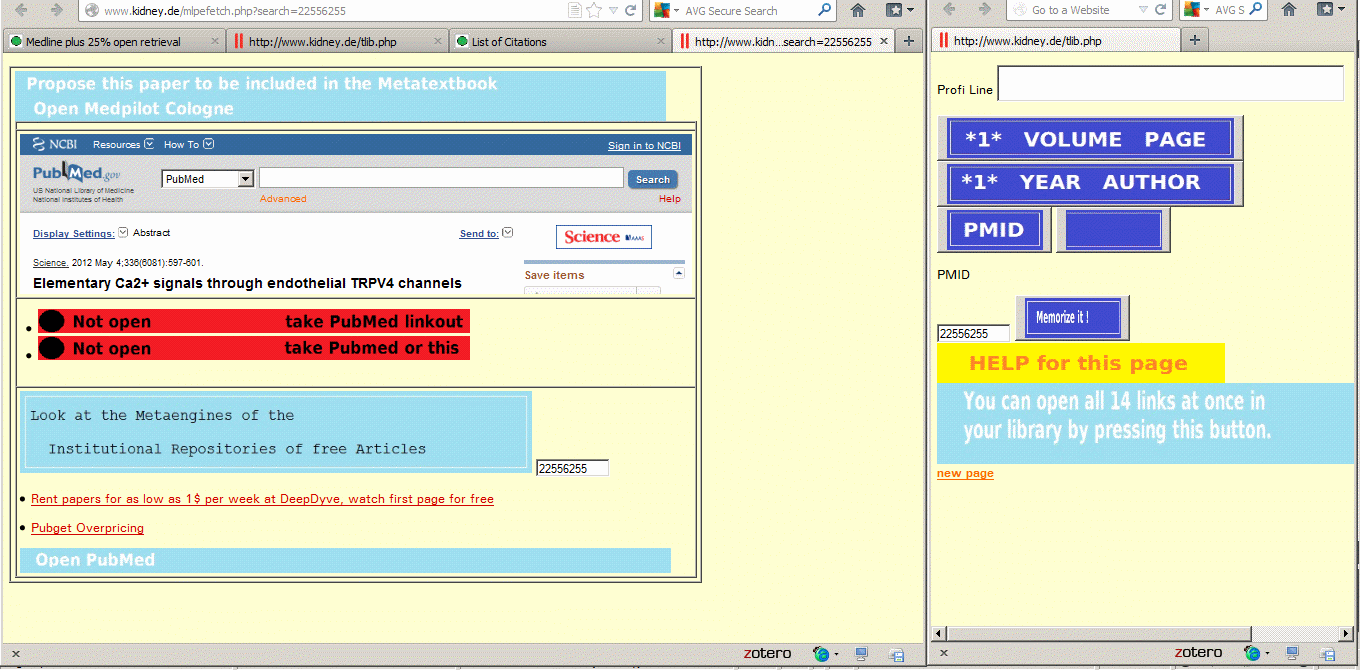
ziehen zunächst die PMID von der common linkout box (steht rechts neben dem button für die Repositories) in das Feld neben dem Memorize it button
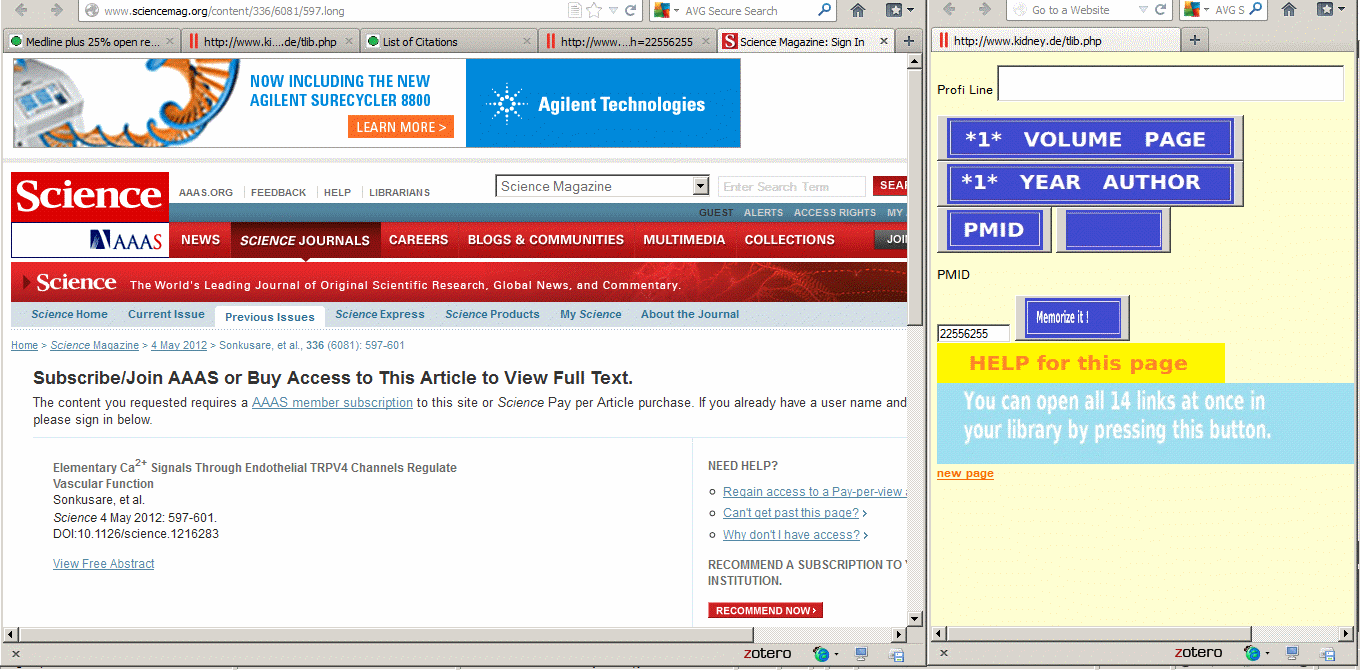
ausgelinkte
Verlagsseite, hier Science
und klicken den Link vom geframten PubMed, welcher die Verlagsseite zum PDF öffnet.
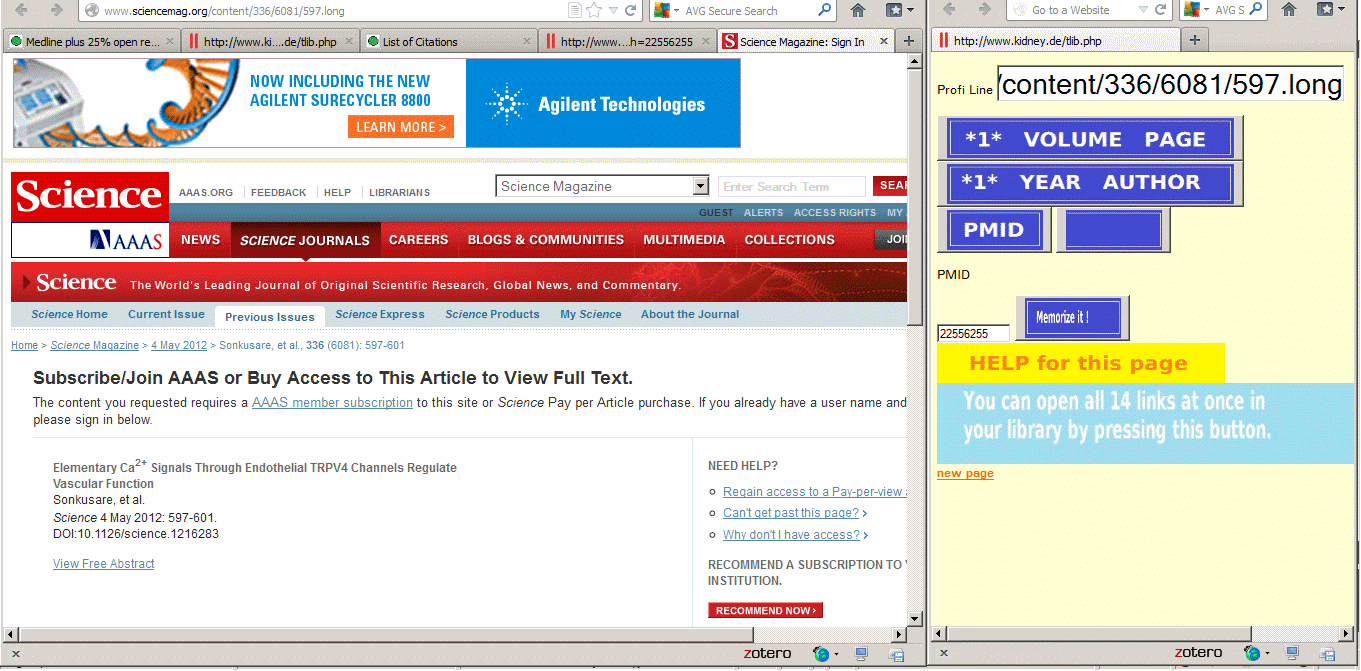
ausgelinkte
Verlagsseite, hier Science
Wir kopieren nun die Adresse dieser Seite bzw.: derjenigen Verlagsseite, auf welcher sich der Link zum Öffnen des PDFs sich befindet in das Profi-Line Feld und drücken auf den Memorize-it Knopf.
ausgelinkte
Verlagsseite, hier Science
Es können pro Datei 14 solcher Links gemerkt werden. Wenn alle Links eingegeben sind, muß die Seite gespeichert werden, verwenden Sie einen relativ systematischen Namen. (Aus Firefox: SAVE PAGE AS...).
Wenn Sie mehr als 14 PDFs besorgen möchten, müssen Sie eine weitere Datei anlegen.
Wenn Sie nun in Ihre Bibliothek gehen wollen, müssen Sie diese .htm Dateien nur noch auf ihren USB Stick kopieren...
Anwender-generierte
Links zu Verlagsseiten, hier Springer
...und dort öffnen: USB Inhaltsverzeichnis öffnen, Datei Rechtsklick Öffnen mit Firefox.
Dann klicken Sie auf das Feld 'Open all 14 Links'. und, wie dargestellt, sollten sich multiple Fenster gleichzeitig öffnen, die zum PDF führen.
Wenn das PDF aufgeht, speichern Sie es auf ihren USB Stick.
Wenn der Link nicht geht, ist das Journal wohl in Ihrer Bibliothek nicht online erhältlich.
Wenn Sie als Nutzer der Kölner 38M ZbMed die Signatur benötigen, so müssen Sie nur ermitteln, welcher von den 14 dargestellten Links betroffen ist und dann auf den Link daneben (CLB) klicken, um die CommonLinkoutBox zu öffnen. Von dort können Sie dann den Medpiloten starten, der Ihnen die Signatur nennt.